用深度图对抗 COVID-19
自2019年12月以来,COVID-19冠状病毒在全球的快速传播已导致超过700万感染病例和超过40万人死亡。COVID-19的快速传播表明迫切需要快速有效的药物发现。药物再利用(Drug repurposing)是一种药物发现范式,它将现有药物用于新的治疗适应症。与从头(de novo)药物发现相比,它具有显著减少时间和成本的优势。利用知识图谱进行药物再利用为COVID-19治疗提供了一种有前景的策略。
由亚马逊上海人工智能实验室和AWS深度引擎科学团队的AWS科学家组成的团队,与明尼苏达大学、俄亥俄州立大学和湖南大学的学术合作者一起,创建了药物再利用知识图谱(DRKG)和一套可用于优先筛选药物进行再利用研究的机器学习工具。DRKG和这些机器学习工具已在GitHub上开源,以帮助研究人员对COVID-19和其他疾病(如阿尔茨海默病)进行药物再利用研究。
DRKG是一个全面的生物学知识图谱,关联了人类基因、化合物、生物过程、药物副作用、疾病和症状。DRKG整合、整理并规范化了来自六个公开可用数据库以及从最近与Covid-19相关的出版物中收集的数据。它包含97,238个实体(属于13种实体类型)和5,874,261个三元组(属于107种关系类型)。
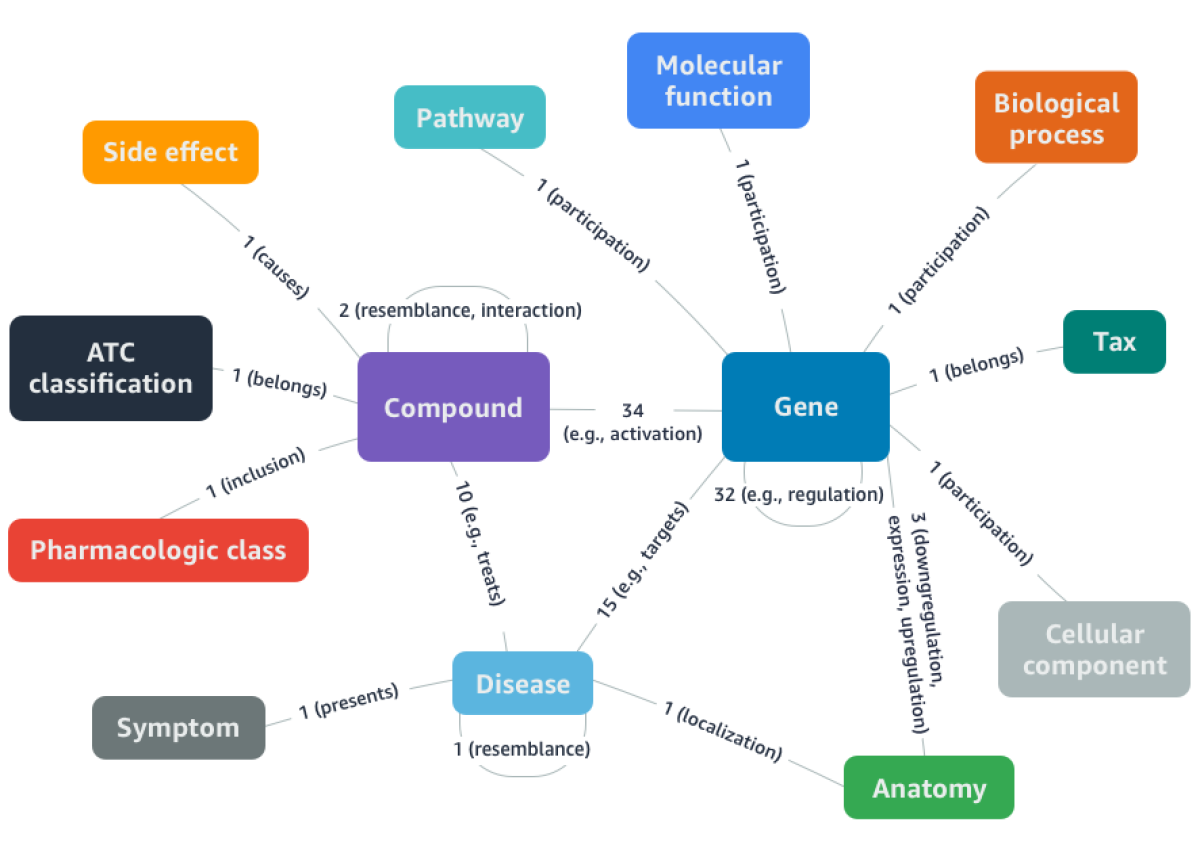
这些机器学习工具使用 DGL-KE 学习 DRKG 中实体和关系的低维嵌入表示。由此产生的嵌入用于预测药物治疗疾病的可能性,或药物与疾病相关蛋白结合的可能性。DGL-KE 是由亚马逊上海人工智能实验室开发的一个高性能、易用且可扩展的软件包,用于学习大规模知识图谱嵌入。该软件包在 Deep Graph Library (DGL) 的基础上实现,开发者可以在 CPU 机器、GPU 机器以及集群上运行 DGL-KE,并支持一系列流行模型,包括 TransE, DistMult, ComplEx, RotatE 等。它可以在具有 8 块 GPU 的 EC2 实例上用 100 分钟训练包含超过 8600 万个节点和 3.38 亿条边的知识图谱,或者在包含 4 台机器的 EC2 集群上用 30 分钟完成训练。
DRKG 的 GitHub 仓库提供了使用 DGL-KE 学习 DRKG 中实体和关系的低维嵌入表示以及使用 DRKG 预训练知识图谱嵌入进行 药物再利用的示例。初步实验结果表明,使用某些机器学习工具进行 COVID-19 药物发现可以识别出多种目前正在临床试验中且排名分数较高的药物。
使用 DGL-KE 学习 DRKG 中实体和关系的低维嵌入表示
DRKG 使用 DGL-KE 学习知识图谱嵌入
步骤 1: 从 此处 下载 DRKG
步骤 2: 按如下方式加载 DRKG。
import sys
sys.path.insert(1, '../utils')
from utils import download_and_extract
download_and_extract()
drkg_file = '../data/drkg/drkg.tsv'
步骤 3: DRKG 数据包中包含一个 drkg.tsv 文件,其中包含知识图谱中的所有三元组。在训练之前,我们按照 0.9:0.05:0.05 的比例随机将数据集划分为训练集、验证集和测试集。
import pandas as pd
import numpy as np
df = pd.read_csv(drkg_file, sep="\t")
triples = df.values.tolist()
seed = np.arange(num_triples)
np.random.shuffle(seed)
train_cnt = int(num_triples * 0.9)
valid_cnt = int(num_triples * 0.05)
train_set = seed[:train_cnt]
train_set = train_set.tolist()
valid_set = seed[train_cnt:train_cnt+valid_cnt].tolist()
test_set = seed[train_cnt+valid_cnt:].tolist()
with open("train/drkg_train.tsv", 'w+') as f:
for idx in train_set:
f.writelines("{}\t{}\t{}\n".format(triples[idx][0], triples[idx][1], triples[idx][2]))
with open("train/drkg_valid.tsv", 'w+') as f:
for idx in valid_set:
f.writelines("{}\t{}\t{}\n".format(triples[idx][0], triples[idx][1], triples[idx][2]))
with open("train/drkg_test.tsv", 'w+') as f:
for idx in test_set:
f.writelines("{}\t{}\t{}\n".format(triples[idx][0], triples[idx][1], triples[idx][2]))
步骤 4: 然后我们直接调用 DGL-KE 提供的命令行工具包来学习 DRKG 中实体和关系的低维嵌入表示。这里我们选择 TransE_l2 模型,并使用 AWS p3.16xlarge 实例进行多 GPU 并行训练。要使用其他 KGE 模型或 AWS 实例,请参考 DGL-KE 的文档。
DGLBACKEND=pytorch dglke_train --dataset DRKG --data_path ./train \
--data_files drkg_train.tsv drkg_valid.tsv drkg_test.tsv \
--format 'raw_udd_hrt' --model_name TransE_l2 --batch_size 2048 \
--neg_sample_size 256 --hidden_dim 400 --gamma 12.0 --lr 0.1 \
--max_step 100000 --log_interval 1000 --batch_size_eval 16 -adv \
--regularization_coef 1.00E-07 --test --num_thread 1 \
--gpu 0 1 2 3 4 5 6 7 --num_proc 8 \
--neg_sample_size_eval 10000 --async_update
步骤 5: 训练完成后,会生成两个文件:1) DRKG_TransE_l2_entity.npy,包含 DRKG 中实体的低维嵌入表示;2) DRKG_TransE_l2_relation.npy,包含 DRKG 中关系的低维嵌入表示。这些嵌入可以用于药物再利用任务。
node_emb = np.load('./ckpts/TransE_l2_DRKG_0/DRKG_TransE_l2_entity.npy')
relation_emb = np.load('./ckpts/TransE_l2_DRKG_0/DRKG_TransE_l2_relation.npy')
完整的示例代码可以在此处找到。
使用预训练的知识图谱嵌入进行 COVID-19 药物再利用——这是来自亚马逊 AWS AI、湖南大学、克利夫兰诊所勒纳基因组医学中心和明尼苏达大学(使用深度学习再利用开放数据发现 COVID-19 疗法)的一项合作工作,他们提出了一种新的 COVID-19 药物再利用方法,结合了知识图谱嵌入和基因集富集分析方法。DRKG 借鉴了类似的想法,并提供了 DRKG 的预训练知识图谱嵌入用于 COVID-19 的药物再利用。
首先,我们将使用 DRKG 知识图谱寻找 COVID-19 药物的任务定义为预测候选药物实体与 COVID-19 相关疾病实体在 'Hetionet::CtD::Compound:Disease' 和 'GNBR::T::Compound:Disease' 关系下的可能连接的任务,即治疗关系。我们从 DRKG 中选择分子量大于 250 的 FDA 批准药物作为候选药物,并使用 DRKG 中与 COVID-19 病毒相关的 34 个实体作为目标实体。然后,我们使用 TransE_L2 算法预测所有可能三元组(药物、治疗、病毒)的连接分数,并对分数进行排序。最后,我们选择得分最高的 100 个连接,并将相应的药物视为再利用药物。详细步骤如下:
步骤 1: 设置目标病毒实体、药物实体和治疗关系。
# COVID-19 related virus entities
COV_disease_list = ['Disease::SARS-CoV2 E','Disease::SARS-CoV2 M', ...]
# treatment relations
treatment = ['Hetionet::CtD::Compound:Disease','GNBR::T::Compound:Disease']
# candidate drugs(provided in infer_drug.tsv along with the whole DRKG dataset)
drug_list = []
with open("./infer_drug.tsv", newline='', encoding='utf-8') as csvfile:
reader = csv.DictReader(csvfile, delimiter='\t', fieldnames=['drug','ids'])
for row_val in reader:
drug_list.append(row_val['drug'])
步骤 2: 获取预训练的 DRKG 知识图谱嵌入。
# Reading pretrained embeddings
entity_emb = np.load('../data/drkg/embed/DRKG_TransE_l2_entity.npy')
rel_emb = np.load('../data/drkg/embed/DRKG_TransE_l2_relation.npy')
drug_ids = th.tensor(drug_ids).long()
disease_ids = th.tensor(disease_ids).long()
treatment_rid = th.tensor(treatment_rid)
drug_emb = th.tensor(entity_emb[drug_ids])
treatment_embs = [th.tensor(rel_emb[rid]) for rid in treatment_rid]
步骤 3: 使用 TransE_L2 算法预测所有可能三元组(药物、治疗、病毒)的连接分数,公式如下:
,其中代表头实体(即药物),代表关系,代表尾实体(即病毒),并且是训练中使用的常数值。
import torch.nn.functional as fn
gamma=12.0
def transE_l2(head, rel, tail):
score = head + rel - tail
return gamma - th.norm(score, p=2, dim=-1)
scores_per_disease = []
dids = []
# predict the connection scores of (Drug, Treatment, Virus) triplets
# for each treatment type and combine them together.
for rid in range(len(treatment_embs)):
treatment_emb=treatment_embs[rid]
for disease_id in disease_ids:
disease_emb = entity_emb[disease_id]
score = fn.logsigmoid(transE_l2(drug_emb, treatment_emb, disease_emb))
scores_per_disease.append(score)
dids.append(drug_ids)
scores = th.cat(scores_per_disease)
步骤 4: 对分数进行排序。
idx = th.flip(th.argsort(scores), dims=[0])
scores = scores[idx].numpy()
dids = dids[idx].numpy()
步骤 5: 获取排名前 100 的再利用药物。
topk=100
_, unique_indices = np.unique(dids, return_index=True)
topk_indices = np.sort(unique_indices)[:topk]
# top-100 drug Ids
proposed_dids = dids[topk_indices]
# top-100 drug scores
proposed_scores = scores[topk_indices]
步骤 6: 在排名前 100 的再利用药物中,有六种药物正在进行临床试验。它们的排名和分数如下:
[0] Ribavirin -0.21416784822940826
[4] Dexamethasone -0.9984006881713867
[8] Colchicine -1.080674648284912
[16] Methylprednisolone -1.1618402004241943
[49] Oseltamivir -1.3885014057159424
[87] Deferoxamine -1.513066053390503
完整的示例代码可以在此处找到。
延伸阅读
09 六月
